高斯混合聚类(GMM)及代码实现
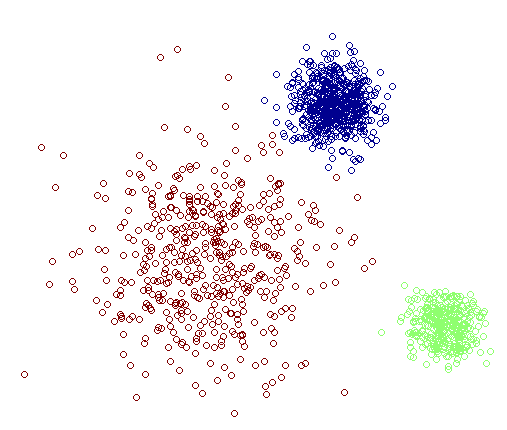
通过学习概率密度函数的Gaussian Mixture Model (GMM) 与 k-means 类似,不过 GMM 除了用在 clustering 上之外,还经常被用于 density estimation。对于二者的区别而言简单地说,k-means 的结果是每个数据点被 assign 到其中某一个 cluster ,而 GMM 则给出这些数据点被 assign 到每个 cluster 的概率。
作为一个流行的算法,GMM 肯定有它自己的一个相当体面的归纳偏执了。其实它的假设非常简单,顾名思义,Gaussian Mixture Model ,就是假设数据服从 Mixture Gaussian Distribution ,换句话说,数据可以看作是从数个 Gaussian Distribution 中生成出来的。实际上,一般在K-means 和 K-medoids 中所用的例子就是由多个Gaussian 分布随机生成的。实际上,从中心极限定理可以看出,Gaussian 分布这个假设其实是比较合理的,除此之外,Gaussian 分布在计算上也有一些很好的性质,所以,虽然我们可以用不同的分布来随意地构造 XX Mixture Model ,但是还是 GMM 最为流行。另外,Mixture Model 本身其实也是可以变得任意复杂的,通过增加 Model 的个数,我们可以任意地逼近任何连续的概率密分布。
每个 GMM 由 K 个 Gaussian 分布组成,每个 Gaussian 称为一个“Component”,这些 Component 线性加成在一起就组成了 GMM 的概率密度函数:
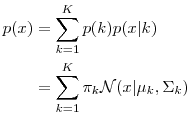
那么如何用 GMM 来做 clustering 呢?其实很简单,假定它们是由 GMM 生成出来的,那么我们只要根据数据推出 GMM 的概率分布来就可以了,然后 GMM 的 K 个 Component 实际上就对应了 K 个 cluster 。根据数据来推算概率密度通常被称作 density estimation 。
现在,假设我们有 N 个数据点,并假设它们服从某个分布(记作 p(x)),现在要确定里面的一些参数的值。 我们的想法是,找到这样一组参数,它所确定的概率分布生成这些给定的数据点的概率最大,而这个概率实际上就等于p(x)之和,我们把这个乘积称作似然函数 (Likelihood Function)。通常单个点的概率都很小,许多很小的数字相乘起来在计算机里很容易造成浮点数下溢,因此我们通常会对其取对数,把乘积变成加和 ,得到 log-likelihood function 。接下来我们只要将这个函数最大化(通常的做法是求导并令导数等于零,然后解方程),亦即找到这样一组参数值,它让似然函数取得最大值,我们就认为这是最合适的参数,这样就完成了参数估计的过程。
下面让我们来看一看 GMM 的 log-likelihood function :

求解最大似然估计过程如下:
1. 估计数据由每个 Component 生成的概率(并不是每个 Component 被选中的概率):对于每个数据 xi 来说,它由第 k 个 Component 生成的概率为
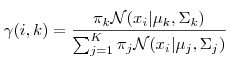
由于式子里的 方差 和 均值 也是需要我们估计的值,我们采用迭代法,在计算时将取上一次迭代所得的值(或者初始值)。
2. 估计每个 Component 的参数:现在我们假设上一步中得到的 r(i,k) 就是正确的“数据 xi 由 Component k 生成的概率”。集中考虑所有的数据点,现在实际上可以看作 Component 生成了所有的 这些点。由于每个 Component 都是一个标准的 Gaussian 分布,可以很容易分布求出最大似然所对应的参数值:
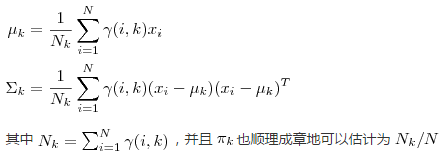
3. 重复迭代前面两步,直到似然函数的值收敛为止。
采用MATLAB自带的kmeansdata数据集进行验证仿真,具体代码如下所示。
%% 导入数据
load('kmeansdata')
%% 初始化混合模型参数
K = 3;
% 随机初始化均值和协方差
means = randn(K,2);
for k = 1:K
covs(:,:,k) = rand*eye(2);
end
priors = repmat(1/K,1,K); % 初始化,假设隐含变量服从先验均匀分布
%% 主算法
MaxIts = 100; % 最大迭代次数
N = size(X,1); % 样本数
q = zeros(N,K); % 后验概率
D = size(X,2); % 维数
cols = {'r','g','b'};
plotpoints = [1:1:10,12:2:30 40 50];
B(1) = -inf;
converged = 0;
it = 0;
tol = 1e-2;
while 1
it = it + 1;
% 把乘除化为对数加减运算,防止乘积结果过于接近于0
for k = 1:K
const = -(D/2)*log(2*pi) - 0.5*log(det(covs(:,:,k)));
Xm = X - repmat(means(k,:),N,1);
temp(:,k) = const - 0.5 * diag(Xm*inv(covs(:,:,k))*Xm'); end
% 计算似然下界
if it > 1
B(it) = sum(sum(q.*log(repmat(priors,N,1))))+ sum(sum(q.*temp)) - sum(sum(q.*log(q)));
if abs(B(it)-B(it-1))<tol
converged = 1;
end
end
if converged == 1 || it > MaxIts
break
end
% 计算每个样本属于第k类的后验概率
temp = temp + repmat(priors,N,1);
q = exp(temp - repmat(max(temp,[],2),1,K));
q(q < 1e-60) = 1e-60;
q(q > (1-(1e-60))) = 1-(1e-60);
q = q./repmat(sum(q,2),1,K); % 更新先验分布
priors = mean(q,1); % 更新均值
for k = 1:K
means(k,:) = sum(X.*repmat(q(:,k),1,D),1)./sum(q(:,k)); end
% 更新方差
for k = 1:K
Xm = X - repmat(means(k,:),N,1);
covs(:,:,k) = (Xm.*repmat(q(:,k),1,D))'*Xm;
covs(:,:,k) = covs(:,:,k)./sum(q(:,k));
end
end
%% plot the data
figure(1);hold on;
plot(X(:,1),X(:,2),'ko')
;for k = 1:K
plot_2D_gauss(means(k,:),covs(:,:,k), -2:0.1:5,-6:0.1:6);
end
ti = sprintf('After %g iterations',it);
title(ti)
%% 绘制似然下界迭代过程图
figure(2);
hold off
plot(2:length(B),B(2:end),'k');
xlabel('Iterations');
ylabel('Bound');
最新评论
推荐文章
作者最新文章
你可能感兴趣的文章
Copyright Disclaimer: The copyright of contents (including texts, images, videos and audios) posted above belong to the User who shared or the third-party website which the User shared from. If you found your copyright have been infringed, please send a DMCA takedown notice to [email protected]. For more detail of the source, please click on the button "Read Original Post" below. For other communications, please send to [email protected].
版权声明:以上内容为用户推荐收藏至CareerEngine平台,其内容(含文字、图片、视频、音频等)及知识版权均属用户或用户转发自的第三方网站,如涉嫌侵权,请通知[email protected]进行信息删除。如需查看信息来源,请点击“查看原文”。如需洽谈其它事宜,请联系[email protected]。
版权声明:以上内容为用户推荐收藏至CareerEngine平台,其内容(含文字、图片、视频、音频等)及知识版权均属用户或用户转发自的第三方网站,如涉嫌侵权,请通知[email protected]进行信息删除。如需查看信息来源,请点击“查看原文”。如需洽谈其它事宜,请联系[email protected]。